- Predstavitev
- Uporaba
- Dostop
- Programski jeziki in knjižnice
- modules
- Nadzornik računskih virov SLURM
- TotalView
- VisIt
- IOzone test
- OpenFOAM
- ppgplasma
- Inkscape
- LaTeX in SVG
- Access
- Analiza, razčlenitev in razhroščevanje
- Ansys
- BIT1
- Cluster Management Utility
- DDT
- Eclipse
- Elmer
- Goldfire Innovator
- MPI
- Mathematica
- NX shadow
- OpenMP
- Platform Managment Console
- Pošta
- R
- Subversion
- UGS NX
- Projekti in dogodki
- Prijava
- Wiki
Main menu
Nahajate se tukaj
VisIt
VisIt je orodje za vizualizacijo podatkov v različnih oblikah. Razvija ga LLNL.
1. 2D izrisi in osnovne lastnosti
- S programom gen_vtk.py, ki je priložen primerom VisIt naredimo podatke mydata0000.vtk. Nato odpremo datoteko v VisItu z File-Open
- Izberemo 2D podatke za izris z Plots-Pseudocolor-Fxy in pritisnemo na Draw
- Spremenimo atribute izrisa z gumbom PlotAtts in spremenimo barvno tabelo tako, da izberemo hot_desaturated in meje, ki jih ta barvna tabela pokriva na [0,3]. Končamo z gumbom Apply
- Rezultat je naslednji:
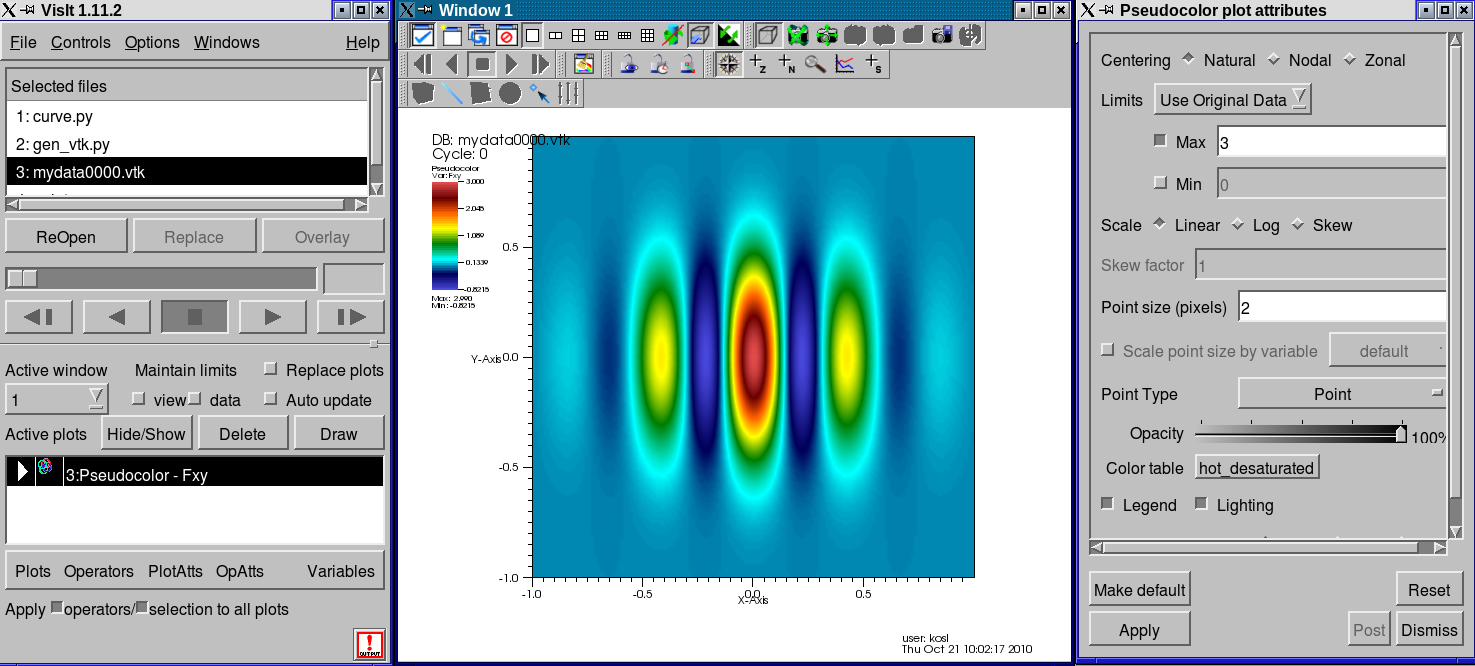
- VisIt omogoča, da kombiniramo prikaze. Contour plot dodamo z eni sami barvi - belo - za vizualizacijo 15 nivojev negativnih vrednosti in odstranimo napise ter na koncu ponovno izvedemo Draw.
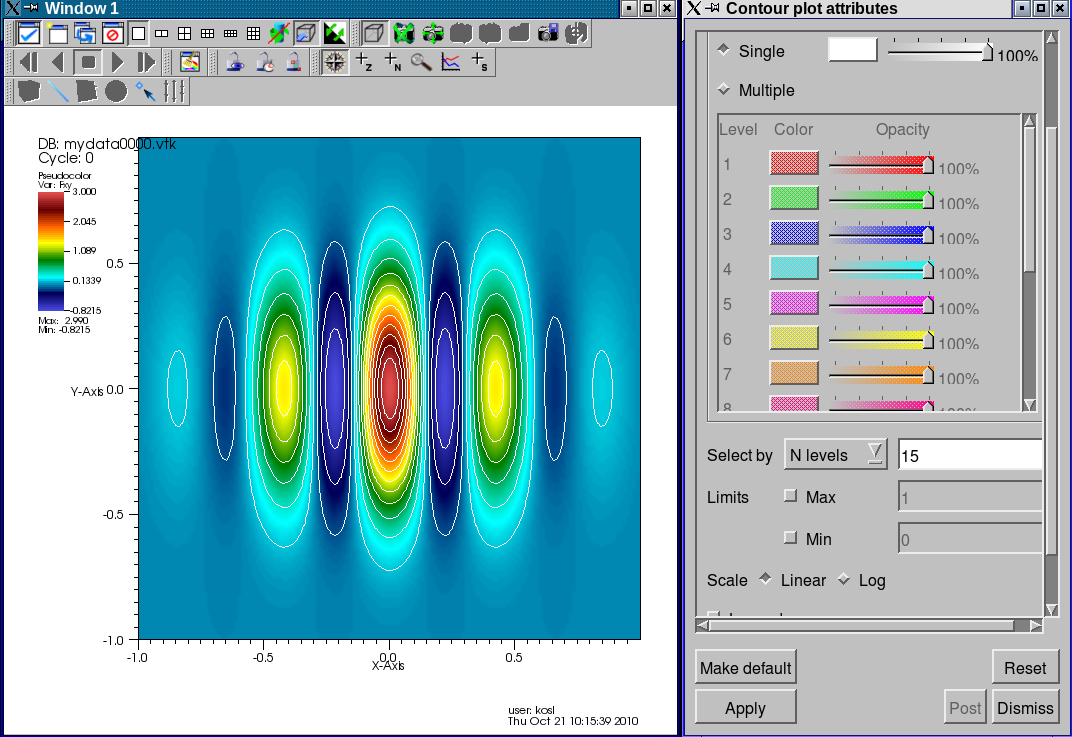
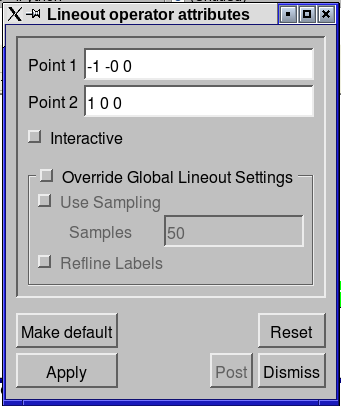
- Če želimo izvleči vrednosti funkcije po črti y=0 kliknemo gumb Lineout, ki je na oknu za prikaz. Kliknemo okoli točke (-1,0) in povlečemo do (1,0). Vrednosti funkcije po tej ročno izbrani črti preseka so izrisani kot 1D krivulja v novem oknu (window 2)
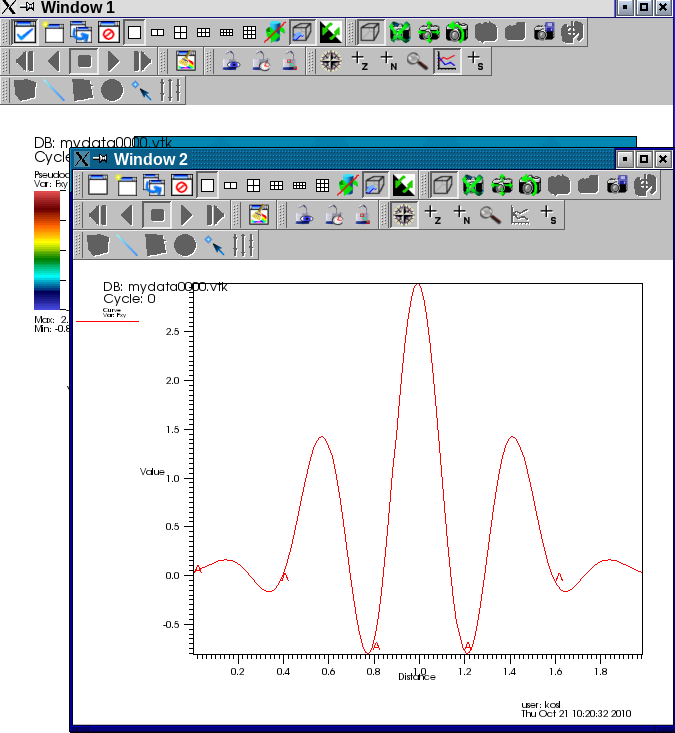
- Za spremembo parametrov v oknu 2 izberemo seznam Window in "active window" in popravimo atribute črtnega izrisa (barva, debelina črte, stil) in odstranimo napis.
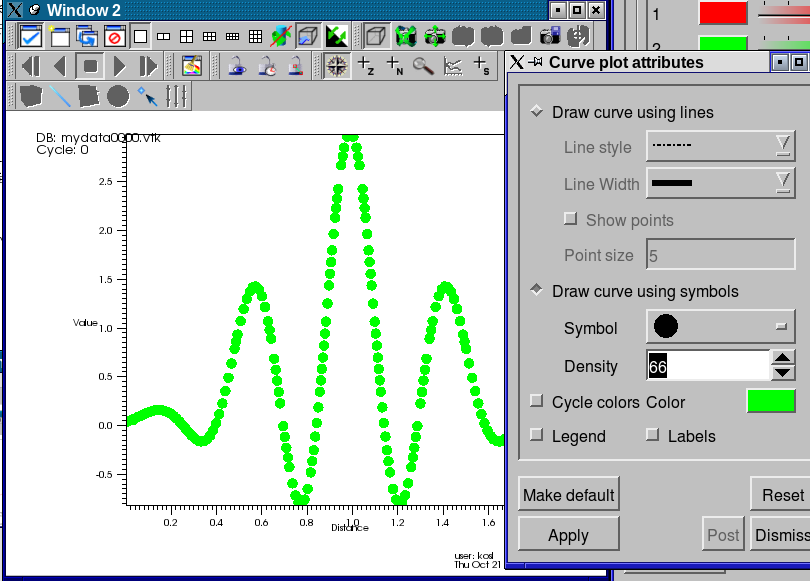
- Z gumbom OpAtts-Lineout lahko natančno nastavimo položaj preseka
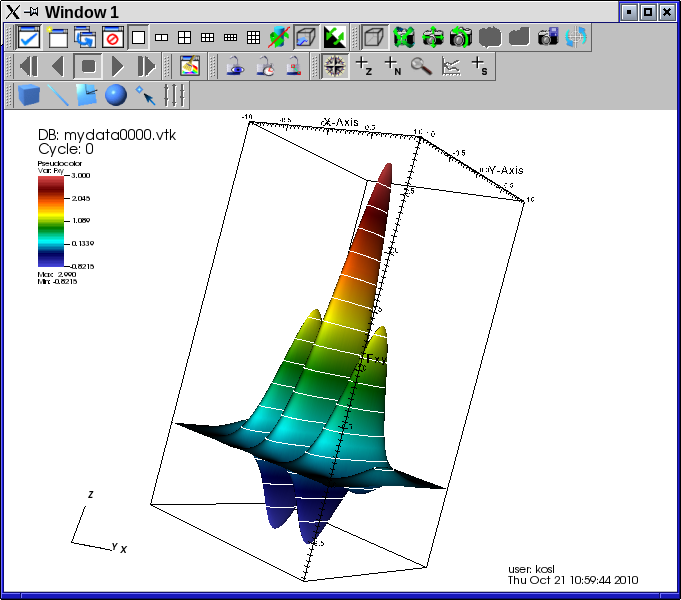
- Na oknu 1 dodamo operator Elevate z višino relativno na XY. S tem transformacijskem operatorju izdelamo 3D prikaz iz 2D podatkov. S klikom in vlečenjem obračamo. Pogled pa prestavljamo z dodatnim tiščanjem tipke CTRL.
- Z uporabo menuja Controls-Annotation spremenimo oznake osi, barve ozadja in ostalo. Preverimo ali antialiasing in specular ligtening izboljša predstavitev.
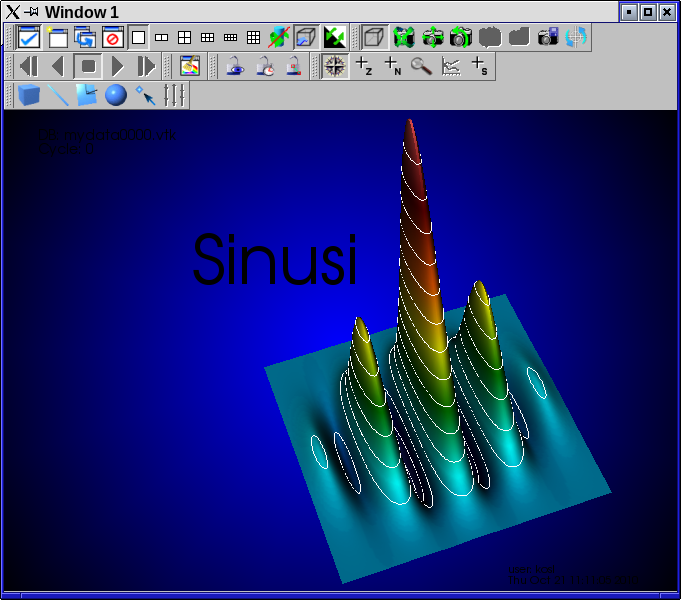
- Na koncu shranimo poglede v dve ločeni rastrski datoteki in v eno skupno datoteko. Izberemo File-Set save options in izberemo format datoteke, ime in postavitev preden izberemo Save.
Izrisi 3D iz zahtevnih binarnih datotek in upravljanje z 3D
Za prikaz bomo najprej tvorili podatke v datoteke tipa SILO z programom v C-ju. Naslednja izvorna koda to naredi s tem da jo prevedemo skupaj s potrebnimi knjižnicami in ukazom
gcc gen_silo_data.c -o gen_silo_data -I /pot-do/silo/local/include -L /pot-do-silo/public/local/lib -lsiloh5 -lhdf5
#include <stdio.h>
#include <stdlib.h>
#include <math.h>
#include <string.h>
#include <silo.h>
#define NBDIM 3
#define NBVAR 1
#define SIZENAME 16
#define SIZEDEF 256
#define NX 32
#define NY 32
#define NZ 32
#define NBSTEP 60
int main()
{
char fileOut[64];
DBfile *dbfile = NULL;
int nxmax[NBDIM]={NX,NY,NZ}; //nb points in each dimensions
double minPhy[NBDIM]={-1.0,-1.0,-1.0}; // Minimum of the physical dimensions
double maxPhy[NBDIM]={1.0,1.0,1.0}; // Maximum of the physical dimensions
double *coord[NBDIM]; // Pointer to the structured mesh
double dx[NBDIM]; //mesh step in each dimension
char* varnames[NBVAR], *vardefs[NBVAR];
int vartypes[NBVAR];
double* f1D, *f2Dx, *f2Dy, *f3D;
double x,y,z;
double t, dt;
int i,j, k, step;
int off0, off1;
off0 = nxmax[0];
off1 = off0*nxmax[1];
//Memory allocation
f1D = (double*) malloc(nxmax[0]*sizeof(double));
f2Dx = (double*) malloc(nxmax[0]*nxmax[1]*sizeof(double));
f2Dy = (double*) malloc(nxmax[0]*nxmax[1]*sizeof(double));
f3D = (double*) malloc(nxmax[0]*nxmax[1]*nxmax[2]*sizeof(double));
//Structured mesh construction
for(i=0; i < NBDIM; ++i)
{
dx[i] = (maxPhy[i]-minPhy[i])/nxmax[i];
coord[i] = (double*) malloc(nxmax[i]*sizeof(double));
for(j = 0; j < nxmax[i]; ++j)
coord[i][j] = minPhy[i] + j*dx[i];
}
for(i=0; i < NBVAR; ++i)
{
varnames[i] = malloc(SIZENAME*sizeof(char));
vardefs[i] = malloc(SIZEDEF*sizeof(char));
}
//Time loop
dt = 1.0/NBSTEP ;
for(step=0; step < NBSTEP; ++step)
{
t=step*dt;
//3D Data evaluation
for(k=0; k < nxmax[2]; ++k)
{
z=minPhy[2] + k*dx[2];
for(j=0; j < nxmax[1]; ++j)
{
y=minPhy[1] + j*dx[1];
for(i=0; i < nxmax[0]; ++i)
{
x=minPhy[0] + i*dx[0];
f3D[i + off0*j + off1*k] = exp(-(x*x*4.0 + y*y*8.0))*(1.0 + 0.3*cos(4.0*M_PI*(z/(maxPhy[2]-minPhy[2]) - t)));
}
}
}
//1D Data extraction from the 3D data
for(k=0; k < nxmax[2]; ++k)
f1D[k] = f3D[nxmax[0]/2 + off1*k];
//2D data evaluation
for(j=0; j < nxmax[1]; ++j)
{
y=minPhy[1] + j*dx[1];
for(i=0; i < nxmax[0]; ++i)
{
x=minPhy[0] + i*dx[0];
f2Dx[i + off0*j] = exp(-(x*x*4.0 + y*y*8.0))*cos(4.0*M_PI*t);
f2Dy[i + off0*j] = exp(-(x*x*4.0 + y*y*8.0))*sin(4.0*M_PI*t);
}
}
//Data export
sprintf(fileOut, "out_%d.silo", step);
dbfile = DBCreate(fileOut, DB_CLOBBER, DB_LOCAL, "file generated by exportSilo.c", DB_HDF5);
if(dbfile == NULL)
{
fprintf(stderr, "Could not create Silo file!\n");
exit(1);
}
//Export 3D mesh and data
DBPutQuadmesh(dbfile, "meshxyz", NULL, (float**) coord, nxmax, 3, DB_DOUBLE, DB_COLLINEAR, NULL); //casting in float** to avoid a warning
DBPutQuadvar1(dbfile, "f3D", "meshxyz", (float*) f3D, nxmax, 3, NULL, 0, DB_DOUBLE, DB_NODECENT, NULL); //casting in float* to avoid a warning
//Export 1D data
DBPutCurve (dbfile, "f1D", coord[0], f1D, DB_DOUBLE, nxmax[0], NULL);
//Export 2D mesh and data
DBPutQuadmesh(dbfile, "meshxy", NULL, (float**) coord, nxmax, 2, DB_DOUBLE, DB_COLLINEAR, NULL); //casting in float** to avoid a warning
DBPutQuadvar1(dbfile, "f2Dx", "meshxy", (float*) f2Dx, nxmax, 2, NULL, 0, DB_DOUBLE, DB_NODECENT, NULL); //casting in float* to avoid a warning
DBPutQuadvar1(dbfile, "f2Dy", "meshxy", (float*) f2Dy, nxmax, 2, NULL, 0, DB_DOUBLE, DB_NODECENT, NULL); //casting in float* to avoid a warning
//Definition of a vector data from the 2 previous scalar data
strcpy(varnames[0], "f2D");
strcpy(vardefs[0], "{f2Dx,f2Dy}");
vartypes[0] = DB_VARTYPE_VECTOR;
DBPutDefvars(dbfile, "myVars", NBVAR, varnames, vartypes, vardefs, NULL);
DBClose(dbfile);
}
//Memory free
free(f1D);
free(f2Dx);
free(f2Dy);
free(f3D);
for(i=0; i < NBVAR; ++i)
{
free(varnames[i]);
free(vardefs[i]);
}
}
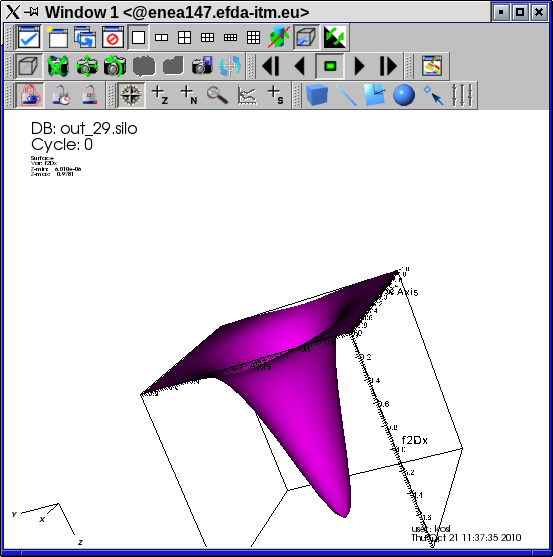
- Po zagonu programa se ustvari množica oštevilčenih SILO datotek, ki vse predstavljajo rezultate ob določenem času. VisIt te datoteke obravnava kot skupno podatkovno bazo, ki jo lahko opremo in opazujemo po časovni osi kot animacijo. Odpremo datoteko v VisIt in dodamo Surface plot za spremenljivko f2Dx. Izrišemo in opazujemo, kaj se dogaja pri animaciji. Nastaviti je potrebno še atribute izrisa in nastaviti konstantne min in max vrednoste ter zakleniti prikaz z gumbom Lock view na oknu prikaza.
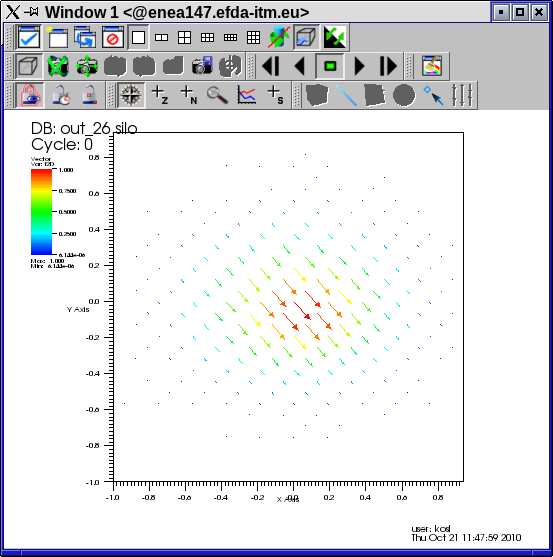
- Izbrišemo trenutni prikaz in naredimo Vector plo za spremenljivko f2D in poženemo animacijo.
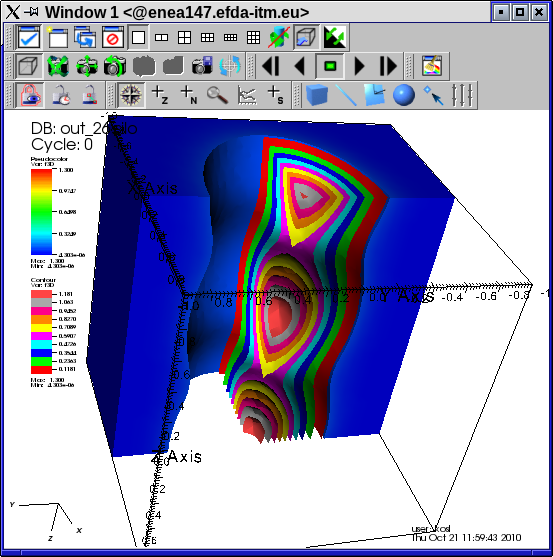
- Izbrišemo trenutni izris in narišemo Pseudocolors in Contour za spremenljivko f3D. Nato dodamo različne operatorje za vsak izris. Izključimo še Apply operators to all plot box. Nato
- dodamo Isovolume k prvem izrisu z min vrednostjo 0.1 in izrez Clip z eno ravnino pravokotno na os x
- dodamo izrez Clip z dvema ravninama definirana z normalama po osi y in z
- Odpremo novo okno tako da liknemo na 1x2 window layout v prikazu prvega okna. V oknu 2 izrišemo Volume plot za spremenljivko f3D. Spremenimo atribute in damo visoko prozornost (Opacity) na rdeči del v barvni tabeli in srednjo prozornost za svetlo modri del. Poskusimo še vklopiti in izklopiti osvetlitev in povečati število vzorcev (postopoma, saj zahteve veliko časa za računanje).
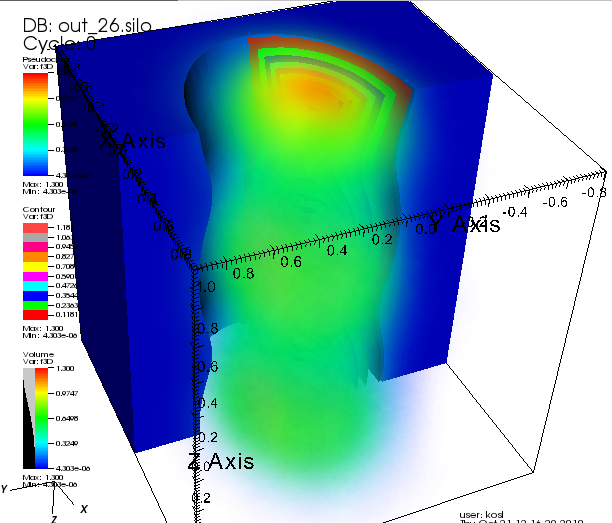
- Če izberemo lock view bo premikanje v enem oknu premikalo tudi drugo okno.
- V oknu 1 izbrišemo prejšnje operatorje in izbrišemo Contour plot. nato dodamo operator Isosurface na ostanek izrisa. Dodamo še en Pseudocolor izris z zmanjšano prozornostjo in Slice operator skupo na oba izrisa ter izrišemo z Draw.
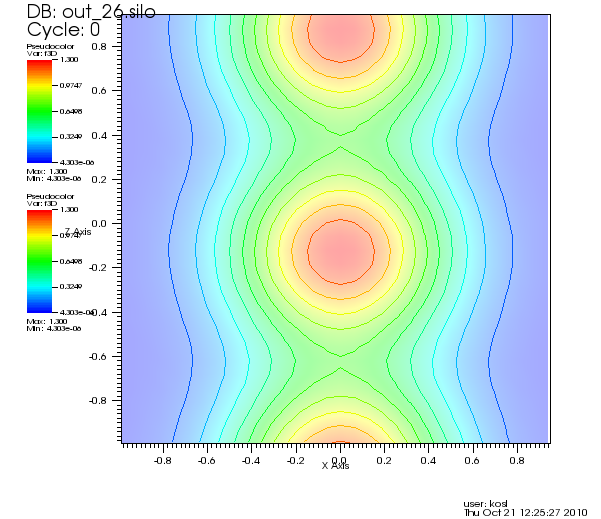
- Na koncu še shranimo sejo z File - Save Session
- Prijavi se za komentar
